卫星图像植被健康分析
本文介绍的使用 Python 进行 NDVI 分析提供了一种通过卫星图像监测植被健康和密度的强大工具。

人类视觉只能感知电磁波谱的一小部分,即“可见光”,这限制了我们对周围环境的狭小视野。然而,现代遥感技术通过使用多光谱图像,将范围远远超出了可见光,多光谱图像可以捕捉到我们眼睛看不见的一系列波长。
卫星或无人机上的传感器测量光谱各个部分或“波段”的反射能量,使每个波段都能显示独特的细节。由于不同的物质(如植被和水)以独特的方式反射能量,多光谱和高光谱图像可以识别各种环境特征。

1、了解 NDVI
归一化差异植被指数 (NDVI) 是一种使用传感器数据测量植被健康和密度的指标。它在以下领域特别有价值:
- 农业监测和作物健康评估
- 森林覆盖率变化检测
- 城市绿地管理
- 干旱监测
- 气候变化影响研究
2、如何计算 NDVI?
NDVI 是根据红光和近红外波段的光谱数据计算得出的,通常来自卫星等远程传感器。它使用一个简单的公式来识别给定图像中植被的存在,计算为红光 (R) 和近红外 (NIR) 值之间的比率:
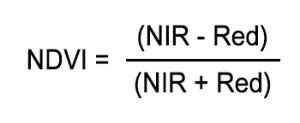
NDVI 的范围始终为 -1 到 +1。但每种土地覆盖类型都没有明确的界限。例如,当 NDVI 值为负时,很可能是水。另一方面,如果 NDVI 值接近 +1,则很有可能是茂密的绿叶。但当 NDVI 接近零时,很可能没有绿叶,甚至可能是城市化区域。

3、使用 Python 计算 NDVI
现在,我们将逐步介绍计算和可视化 NDVI 的每个步骤。
导入库:
# Import libraries
import numpy as np
import rasterio
import matplotlib.pyplot as plt
from matplotlib.colors import LinearSegmentedColormap首先,我们导入以下库:
numpy用于处理数值计算,rasterio用于读取、写入和处理栅格数据(地理空间图像),matplotlib.pyplot用于数据可视化,LinearSegmentedColormap用于定义自定义颜色图以显示 NDVI 值。
加载并准备数据:
# Load and prepare data
b4 = rasterio.open('LC09_L1TP_227082_20231015_20231015_02_T1_B4.TIF').read(1)
b5 = rasterio.open('LC09_L1TP_227082_20231015_20231015_02_T1_B5.TIF').read(1)
red = b4.astype('float64')
nir = b5.astype('float64')接下来,我们从 Landsat 9 图像中加载红色和近红外波段(波段 4 和波段 5)。 astype('float64') 将像素值转换为浮点数,以便进行准确的 NDVI 计算。
定义 NDVI 计算函数:
# Define the NDVI calculation function
def calculate_ndvi(nir_band, red_band):
"""Calculate NDVI from NIR and Red bands"""
ndvi = (nir_band - red_band) / (nir_band + red_band + 1e-10)
ndvi = np.clip(ndvi, -1, 1)
return ndvi
# Calculate NDVI
ndvi_result = calculate_ndvi(nir, red)此函数通过应用 NDVI 公式计算 NDVI。 np.clip(ndvi, -1, 1) 确保 NDVI 值保持在 -1 到 1 的预期范围内,处理任何浮点错误。
将 NDVI 保存为 GeoTIFF:
# Save NDVI as GeoTIFF
with rasterio.open('LC09_L1TP_227082_20231015_20231015_02_T1_B4.TIF') as src:
profile = src.profile
profile.update(
dtype=rasterio.float32,
count=1,
compress='lzw',
nodata=None
)
with rasterio.open('ndvi_result.tif', 'w', **profile) as dst:
dst.write(ndvi_result.astype(rasterio.float32), 1)然后,我们将计算出的 NDVI 数组保存为 GeoTIFF 文件,使用与原始文件相同的元数据进行设置,并将数据类型更新为 float32 以确保准确性。
创建并显示 NDVI 地图:
# Create and display NDVI map
plt.figure(figsize=(12, 10))
colors = ['red', 'yellow', 'lightgreen', 'darkgreen']
ndvi_cmap = LinearSegmentedColormap.from_list("custom", colors, N=256)
im = plt.imshow(ndvi_result, cmap=ndvi_cmap, vmin=-1, vmax=1)
plt.colorbar(im, label='NDVI Value')
plt.title('NDVI Map', pad=20)
plt.axis('off')
plt.show()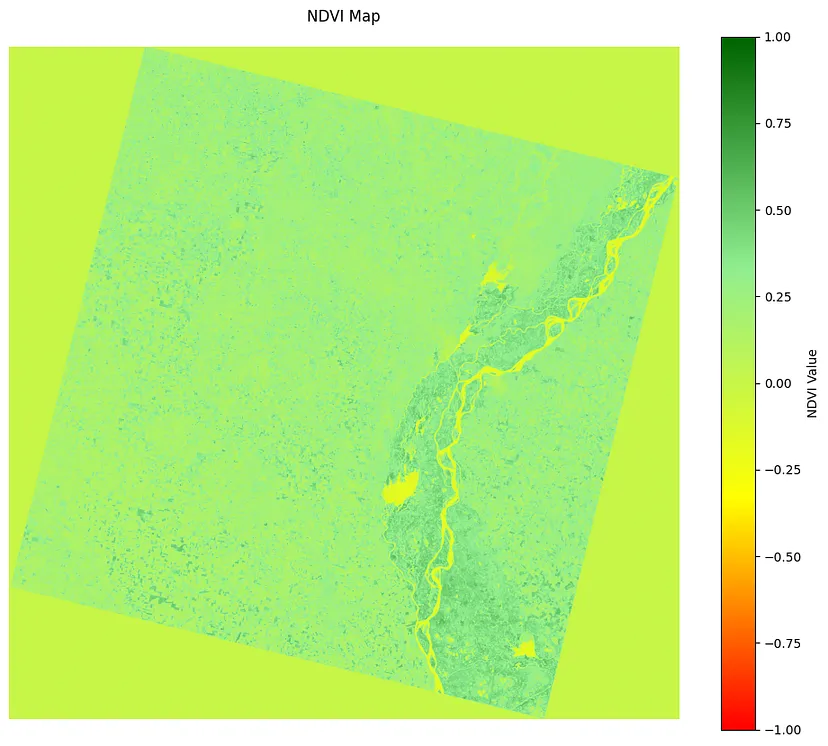
这样,我们创建了一个 NDVI 颜色图,其渐变色从红色(低 NDVI)到深绿色(高 NDVI),显示彩色 NDVI 地图。
计算基本统计数据:
# Calculate basic statistics
valid_ndvi = ndvi_result[~np.isnan(ndvi_result)]
stats = {
'Mean': np.mean(valid_ndvi),
'Median': np.median(valid_ndvi),
'Std Dev': np.std(valid_ndvi),
'Min': np.min(valid_ndvi),
'Max': np.max(valid_ndvi)
}
for stat, value in stats.items():
print(f"{stat}: {value:.3f}")
我们还可以计算 NDVI 值的关键统计数据,排除任何无效值 (NaN),以获得植被健康的清晰的摘要。
创建并显示直方图:
# Create and display a histogram
plt.figure(figsize=(12, 6))
plt.hist(valid_ndvi.flatten(), bins=100, color='darkgreen', alpha=0.7)
plt.title('NDVI Distribution')
plt.xlabel('NDVI Value')
plt.ylabel('Frequency')
plt.grid(True, alpha=0.3)
# Add statistics to plot
stats_text = '\n'.join([f'{k}: {v:.3f}' for k, v in stats.items()])
plt.text(0.95, 0.95, stats_text,
transform=plt.gca().transAxes,
verticalalignment='top',
horizontalalignment='right',
bbox=dict(boxstyle='round', facecolor='white', alpha=0.8))
plt.show()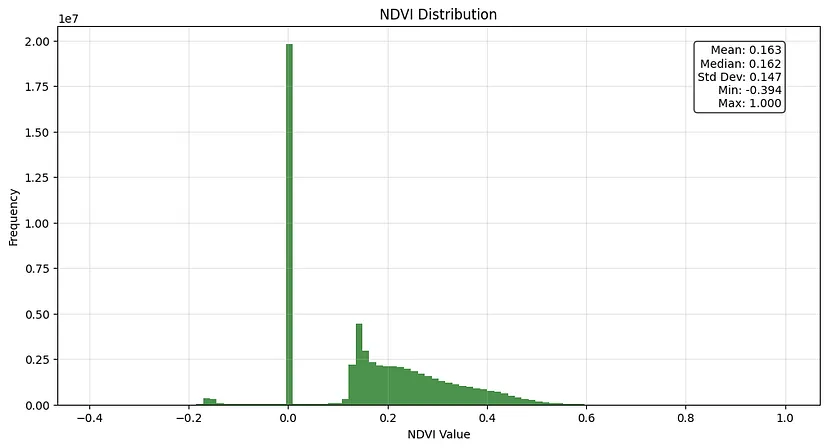
我们可以绘制 NDVI 值的直方图,以直观地分析其分布,并在图中显示关键统计数据。
为高于阈值的区域创建可视化和统计信息:
# Create visualization and statistics for areas above threshold
def analyze_ndvi_threshold(ndvi_data, threshold):
"""
Plot NDVI areas above threshold and display statistics
Parameters:
ndvi_data: numpy array of NDVI values
threshold: NDVI value above which to show areas
"""
plt.figure(figsize=(12, 10))
# Create mask for values above threshold
high_ndvi_mask = ndvi_data > threshold
# Create masked array where values below threshold are transparent
masked_ndvi = np.ma.masked_where(~high_ndvi_mask, ndvi_data)
# Create colormap for high NDVI values
colors_high = ['lightgreen', 'green', 'darkgreen']
high_ndvi_cmap = LinearSegmentedColormap.from_list("custom_high", colors_high, N=256)
# Plot with white background
plt.imshow(np.zeros_like(ndvi_data), cmap='Greys', alpha=0.1)
plt.imshow(masked_ndvi, cmap=high_ndvi_cmap, vmin=threshold, vmax=1)
# Add colorbar
cbar = plt.colorbar(label='NDVI Value')
cbar.set_ticks(np.linspace(threshold, 1, 5))
# Calculate statistics for areas above threshold
high_ndvi_values = ndvi_data[high_ndvi_mask]
total_pixels = ndvi_data.size
pixels_above = high_ndvi_values.size
percentage_above = (pixels_above / total_pixels) * 100
stats = {
'Total pixels': total_pixels,
'Pixels above threshold': pixels_above,
'Percentage above threshold': f"{percentage_above:.1f}%",
'Mean NDVI': f"{np.mean(high_ndvi_values):.3f}",
'Median NDVI': f"{np.median(high_ndvi_values):.3f}",
'Max NDVI': f"{np.max(high_ndvi_values):.3f}",
'Min NDVI': f"{np.min(high_ndvi_values):.3f}",
'Std Dev': f"{np.std(high_ndvi_values):.3f}"
}
# Add title
plt.title(f'Areas with NDVI > {threshold}\n'
f'({percentage_above:.1f}% of total area)',
pad=20)
plt.axis('off')
plt.show()
# Print statistics
print(f"\nStatistics for NDVI > {threshold}:")
print("-" * 40)
for key, value in stats.items():
print(f"{key + ':':25} {value}")
return high_ndvi_mask, stats
# Cell 2: Create visualizations and get statistics for different thresholds
thresholds = [0.1, 0.3, 0.5]
# Dictionary to store statistics for all thresholds
all_stats = {}
for threshold in thresholds:
mask, stats = analyze_ndvi_threshold(ndvi_result, threshold)
all_stats[threshold] = stats
# Cell 3: Optional - Create comparative summary table
import pandas as pd
# Convert statistics to DataFrame for easy comparison
df_stats = pd.DataFrame(all_stats).round(3)
print("\nComparative Summary of All Thresholds:")
print("-" * 60)
print(df_stats)

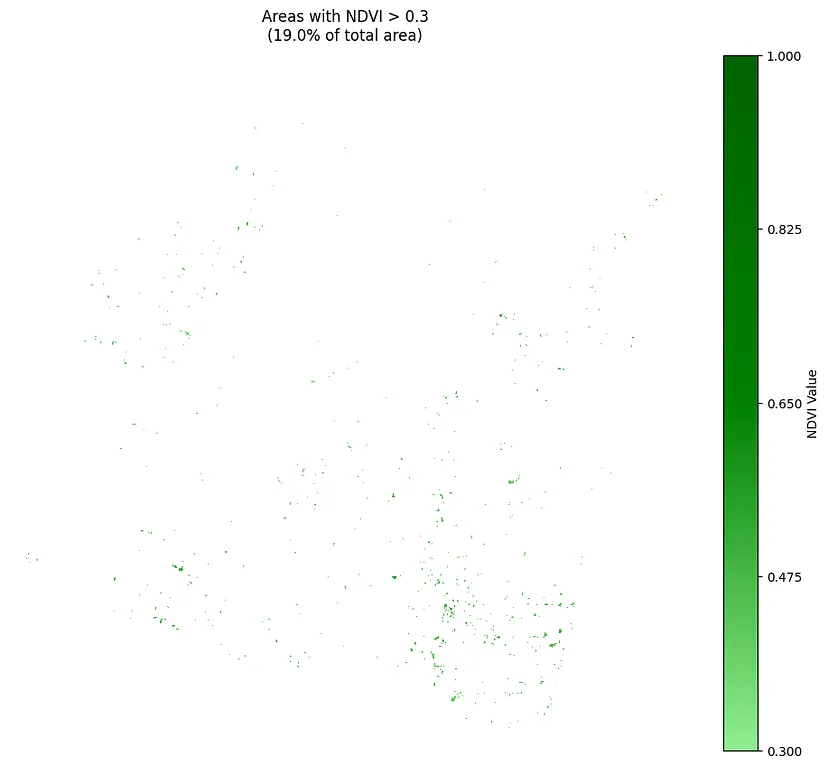

最后,我们可以构建一个函数来分析高于指定阈值(0.1、0.3 和 0.5)的 NDVI 区域,并为这些区域生成单独和比较统计数据。
4、结束语
使用 Python 进行 NDVI 分析提供了一种通过卫星图像监测植被健康和密度的强大工具。此处介绍的工作流程可作为更复杂的环境监测应用的基础。
对于那些希望在此基础上进行构建的人,请考虑探索其他植被指数,如增强植被指数 (EVI)、土壤调整植被指数 (SAVI) 或归一化差异水指数 (NDWI)。此外,您可以实施时间序列分析来跟踪季节变化或将此分析集成到自动监测系统中。
原文链接:Computer Vision | Analyzing Satellite Images using Python
汇智网翻译整理,转载请标明出处
